Metagenómica viral en ambientes extremos
Los virus son las entidades biológicas más abundantes en la biosfera y tienen la capacidad de infectar bacterias, arqueas y eucariotas. Se estima que el viroma es al menos diez veces más abundante que el microbioma con 107 virus por mililitro y 109 partículas virales por gramo en aguas marinas y sedimentos o suelos, respectivamente. Los virus representan una diversidad genética en gran parte inexplorada, y tienen un papel importante en la plasticidad genómica de sus huéspedes. Además, también juegan un papel importante en la dinámica de las poblaciones microbianas.
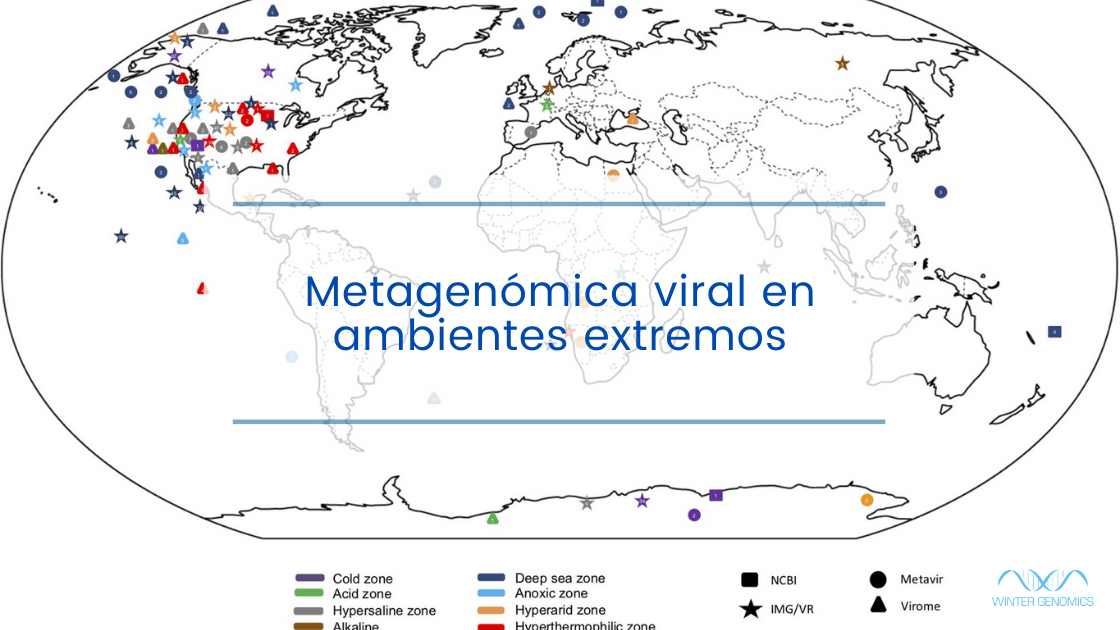
En los últimos años, los enfoques metagenómicos han ganado una popularidad creciente en el estudio de viromas ambientales, ofreciendo la posibilidad de ampliar nuestro conocimiento relacionado tanto con la diversidad de virus como con su caracterización funcional. Los ambientes extremos representan una fuente interesante tanto de microbiota como de su viroma debido a sus particulares condiciones fisicoquímicas, como temperaturas muy altas o muy bajas y presiones hidrostáticas> 1 atm, entre otras.
A pesar de que se han logrado algunos avances en nuestra comprensión de la ecología de la microbiota en estos hábitats, pocos estudios metagenómicos han descrito los viromas presentes en ecosistemas extremos. Por lo tanto, se han realizado avances limitados en nuestra comprensión de la estructura de la comunidad de virus en ecosistemas extremófilos, así como en su potencial biotecnológico.
La metagenómica es un enfoque poderoso para estudiar la estructura viroma de ambientes extremos y sus posibles aplicaciones biotecnológicas en varios campos. Sin embargo, a pesar de su potencial, se han realizado pocos estudios para caracterizar las comunidades virales en estos entornos. Es necesario superar algunos desafíos metodológicos para garantizar que se puedan obtener muestras enriquecidas en partículas virales, así como aumentar los rendimientos de ácidos nucleicos virales que se pueden aislar.
Referencias:
Front. Microbiol., 18 October 2019 https://doi.org/10.3389/fmicb.2019.02403


