Genoma completo vs Exoma completo
La secuenciación de próxima generación (NGS) es una herramienta invaluable en la investigación genética humana y el diagnóstico clínico. El uso práctico de los métodos NGS se ha incrementado drásticamente con el desarrollo de enfoques de secuenciación dirigida, como la secuenciación del exoma completo (WES) o la secuenciación dirigida de paneles de genes. WES surgió como una alternativa eficiente a la secuenciación del genoma completo (WGS) debido al menor costo de secuenciación y a la simplificación del análisis de variantes y el almacenamiento de datos.
Más del 80% de todas las variantes reportadas en ClinVar, y más del 89% de las variantes reportadas como patógenas, provienen de la parte codificante de proteínas del genoma; este número aumenta al 99% cuando se incluye la proximidad inmediata de CDS. Incluso teniendo en cuenta el sesgo de muestreo, existe un acuerdo general de que la mayoría de las enfermedades hereditarias parecen estar causadas por alteraciones en las regiones codificantes de proteínas del genoma. Dado esto, WES ha dominado los proyectos que caracterizan la variación del genoma humano, así como las aplicaciones clínicas.
El proyecto 1000 Genomes no pudo caracterizar estadísticamente muchas de las variantes raras críticas para el diagnóstico de la enfermedad mendeliana debido a un tamaño de muestra limitado. En un intento por obtener una imagen representativa de la variación de la codificación de proteínas en la población humana, se secuenciaron 6.500 muestras de WES durante el proyecto ESP65006. Cuando el Exome Aggregation Consortium (ExAC) compiló y procesó uniformemente un conjunto de referencia mucho más grande de 60,706 experimentos WES, aumentó drásticamente la precisión de la estimación de la frecuencia alélica (AF) en la población general. Esto llevó a la sorprendente conclusión de que hasta el 90% de las variantes notificadas como causantes de la enfermedad mendeliana en la base de datos de ClinVar se observan con demasiada frecuencia en controles sanos como para causar la enfermedad directamente.
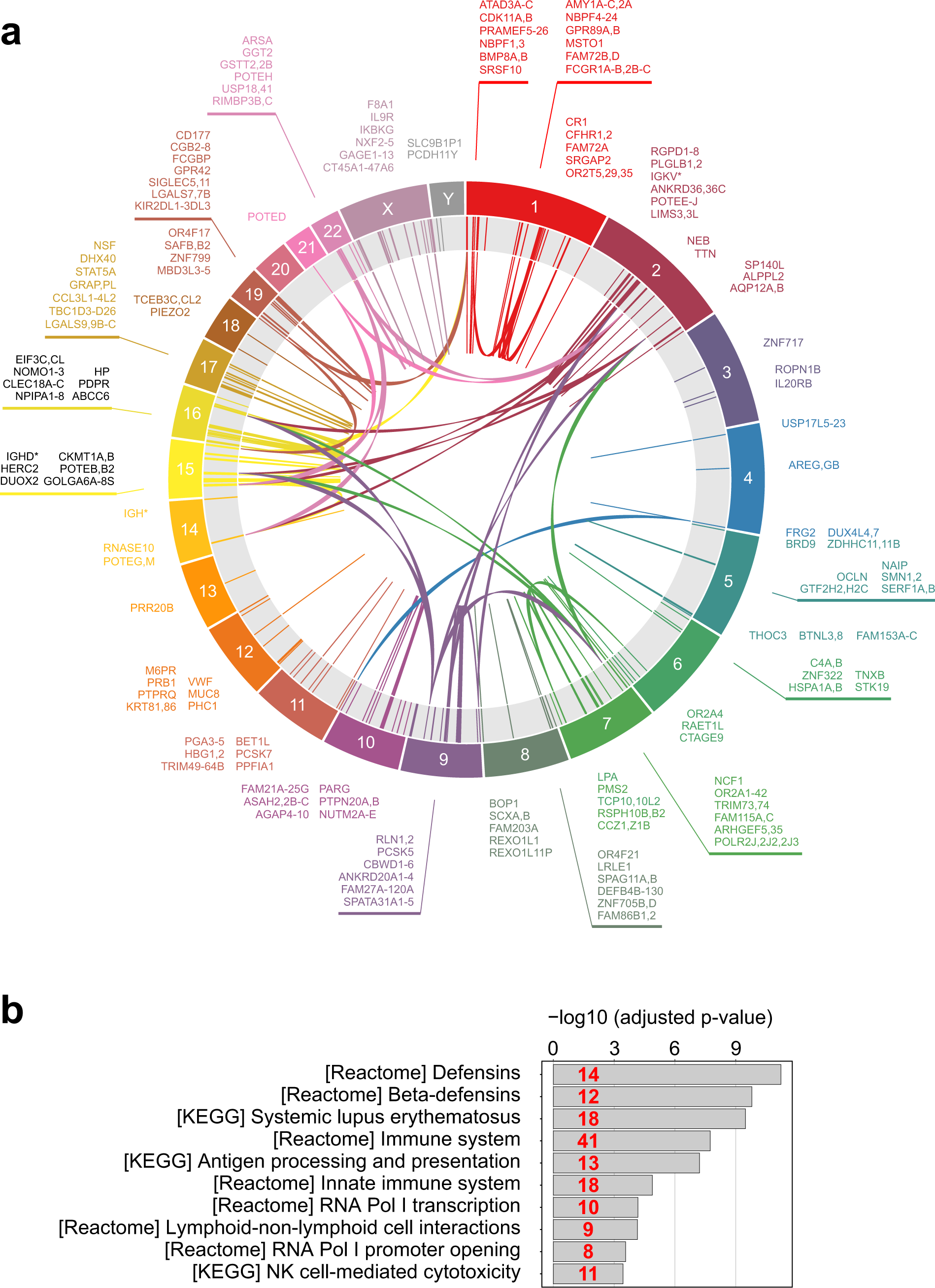
El número de experimentos WES disponibles está aumentando rápidamente, y la última colección de la base de datos de agregación del genoma (gnomAD) incluye 123,136 experimentos WES junto con 15,496 WGS. Un número tan impresionante de individuos perfilados permite una mirada mucho más exhaustiva a la variación del genoma codificante humano, lo que lleva a muchas aplicaciones útiles, como la estimación de la presión selectiva en las regiones codificantes de proteínas.
Las ventajas y la eficacia diagnóstica de WGS y WES se debaten a menudo. WES dominó los proyectos de resecuenciación a gran escala debido a su menor costo y al almacenamiento y procesamiento de datos más fáciles. El rápido desarrollo de métodos de secuenciación de tercera generación y los nuevos kits de secuenciación del exoma predican la necesidad de un marco estadístico robusto que permita una comparación fácil e informativa del rendimiento de los métodos emergentes.
Wright et al. sugieren que WGS es más eficiente que WES solo en un 2% de las tasas de diagnóstico en conjunto. Sin embargo, Barbitoff et al., sugieren que WGS permite una cobertura más eficiente de solo el 1% del exoma en comparación con las mejores plataformas WES, lo que complementa el hecho de que solo una pequeña fracción de las variantes patógenas de ClinVar informadas no son el objetivo de los kits de exoma. Las tasas moderadas de diagnósticos de enfermedades monogénicas basadas en NGS probablemente se explican por la falta de comprensión biológica de la patogenicidad variante. Esto, a su vez, disminuye el papel de los beneficios técnicos de WGS, como la capacidad para identificar numerosas variantes reguladoras en regiones intrónicas e intergénicas. En muchos casos, el reanálisis anual de muestras no diagnosticadas utilizando nuevos datos biológicos mejora la tasa de diagnóstico. Sin duda, el valor de WGS aumentará con una mejor comprensión de la regulación del genoma humano.
Varias líneas de evidencia indican que WES moderno sigue siendo una excelente alternativa a WGS en investigación y aplicaciones clínicas. Además, las tecnologías WES actuales se pueden mejorar aún más de varias maneras: primero, el soporte para tamaños de inserto más largos disminuiría el impacto tanto de la cartografía como de WIE; en segundo lugar, la inclusión de todas las regiones CDS anotadas actualmente para que la cobertura sea más completa; y tercero, un mejor diseño de la sonda y la mejora del proceso de hibridación aliviarían la desigualdad restante resultante del contenido de GC u otros determinantes basados en la secuencia.
Las soluciones WES más recientes (por ejemplo, producidas por Illumina en conjunto con IDT) funcionan sustancialmente mejor que los kits NR o TS analizados en este trabajo, lo que hace que las muestras WES se acerquen a WGS en términos de distribución de cobertura y eventualmente minimizando el diagnóstico. brecha entre los enfoques WES y WGS. Cuando los métodos de tercera generación se adopten más ampliamente, se podrían imaginar métodos combinatorios que se beneficiarían del poder de lectura larga del descubrimiento de variantes complejas, combinado con una alta precisión y rentabilidad de WES.
Referencias:
Barbitoff, Y.A., Polev, D.E., Glotov, A.S. et al. Systematic dissection of biases in whole-exome and whole-genome sequencing reveals major determinants of coding sequence coverage. Sci Rep10, 2057 (2020). https://doi.org/10.1038/s41598-020-59026-y