¿Cómo se realiza el análisis de datos de RNA-Seq?
Los experimentos de RNA-Seq generan un gran volumen de lecturas de secuencias sin procesar, que deben analizarse para interpretar la información. El análisis de datos generalmente requiere una combinación de herramientas de softwares bioinformáticos que varían según el diseño experimental y los objetivos.
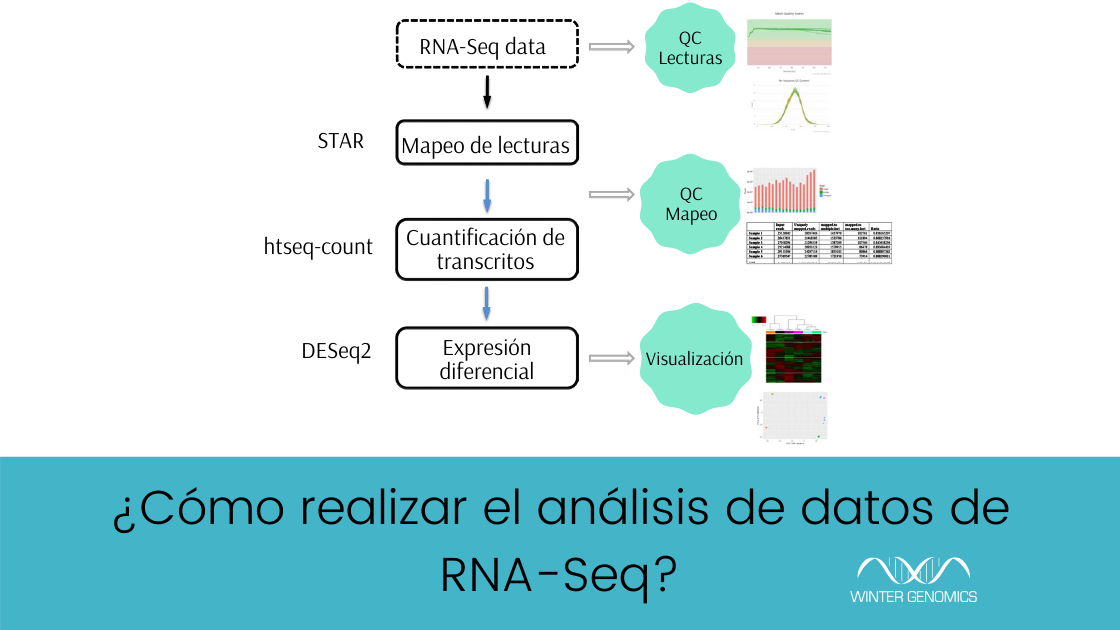
El proceso de análisis de datos de RNA-Seq se puede dividir en:
- Control de calidad
- Alineación y mapeo
- Cuantificación
- Expresión diferencial
Los programas RNA-Seq más populares se ejecutan desde una interfaz de línea de comandos, ya sea en un entorno Unix o dentro del entorno estadístico R / Bioconductor.
¡Aprende a analizar los datos de RNA-Seq!
En este curso teórico-práctico utilizarás las herramientas asociadas con el análisis de datos RNA-Seq mediante línea de comandos. Analizarás los datos crudos, realizarás los filtros de calidad, ensamblaje y anotación de tus datos. Posteriormente se analizarán los datos procesados para conocer la expresión diferencial. No necesitas tener conocimientos previos de programación.
Revisa nuestro curso aquí.
Control de calidad
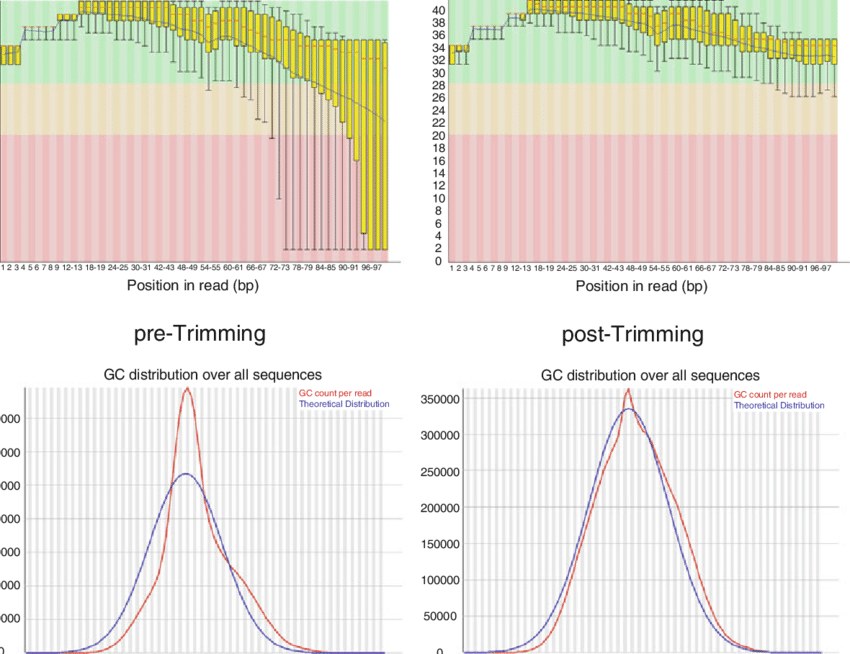
Al terminar el proceso de secuenciación, es necesario estimar la precisión de cada base de la secuencia para los análisis posteriores. Los datos brutos se examinan en busca de puntuaciones de alta calidad para las llamadas de base, el contenido de guanina-citosina coincide con la distribución esperada, la sobrerrepresentación de secuencias particularmente corta (k-mers) y una tasa de duplicación de lecturas inesperadamente altas. Existen varias opciones para el análisis de la calidad, como FastQC. Las secuencias de baja calidad pueden eliminarse recortando y filtrando para continuar con los análisis posteriores.
Alineación
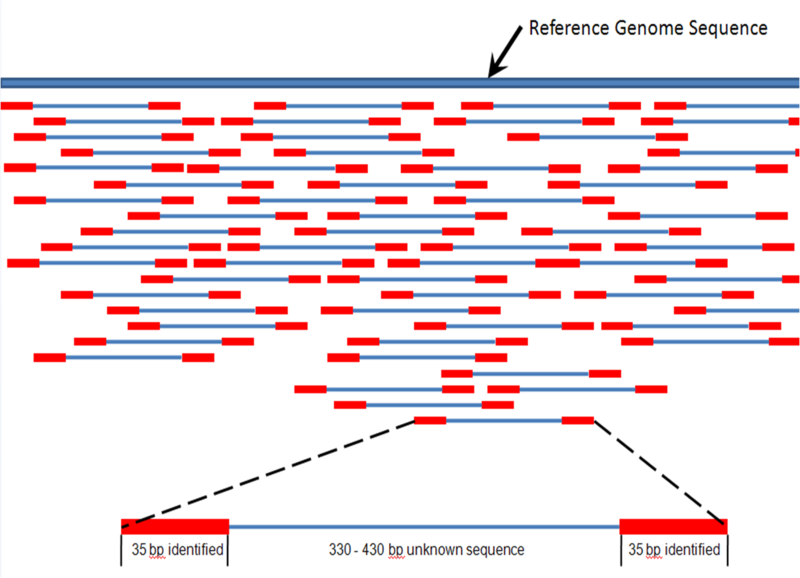
Para conocer la abundancia de secuencias leídas a la expresión de un gen de interés, las secuencias se alinean con un genoma de referencia, o de novo si es que no hay ningún genoma de referencia disponible.
El alineamiento de las secuencias de ARNm a un genoma de referencia requiere un manejo especializado de las secuencias de intrones, que están ausentes del ARNm maduro. La identificación de las uniones de empalme de intrones evita que las lecturas se desalineen a través de las uniones de empalme o se descarten por error, lo que permite alinear más lecturas con el genoma de referencia y mejorar la precisión de las estimaciones de expresión génica. Debido a que la regulación génica puede ocurrir a nivel de isoforma de ARNm, los alineadores también permiten la detección de cambios en la abundancia de isoformas que de otro modo se perderían en un análisis masivo.
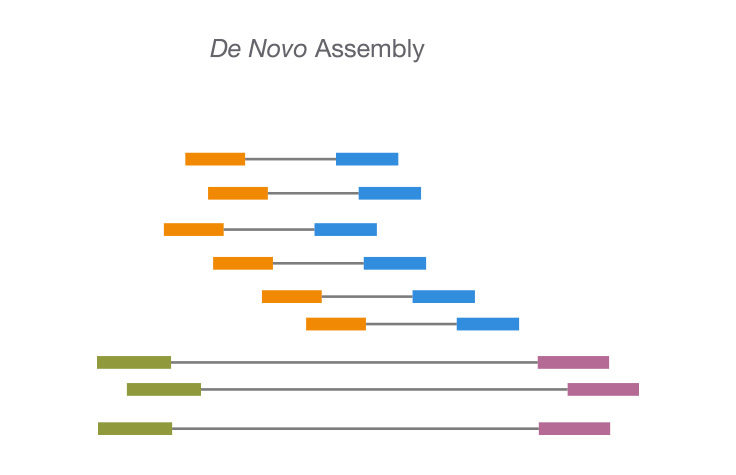
En el ensamblaje de novo, se alinean las lecturas entre sí para construir secuencias de transcripción sin el uso de un genoma de referencia. Los desafíos particulares del ensamblaje de novo son computacionales principalmente, ya que se requieren más recurso en comparación con el ensamblado de referencia. Además, es necesaria la validación adicional de variantes o fragmentos de genes y la anotación adicional de los transcritos ensamblados. transcripciones ensambladas. Una vez realizado el ensamblado de novo, el ensamblaje obtenido se puede utilizar como referencia para los métodos de alineación de secuencias posteriores y el análisis cuantitativo de la expresión génica.
Cuantificación
La cuantificación de las lecturas alineadas se realiza a nivel de gen, exón o transcrito. Los resultados típicos incluyen una tabla de recuentos de lecturas. por ejemplo, para genes en un archivo de formato de característica general. El conteo se puede realizar utilizando softwares como HTSeq o FeatureCounts. La cuantificación a nivel de transcritos requiere métodos estadísticos para estimar la abundancia de las isoformas, utilizando programas como Cufflinks o StringTie.
Kallisto y Salmon son programas que combinan el pseudoalineamiento y la cuantificación en un solo paso con menor carga computacional.

Expresión diferencial
Una vez que se obtienen los datos de conteo, se realiza la expresión diferencial. En este paso se normaliza, modela y analizan estadísticamente los datos. Algunas paqueterías de R comúnmente utilizados son EdgeR y DESeq2. Los resultados finales de estos análisis son listas de genes asociadas por pares para la expresión diferencial entre tratamientos y las estimaciones de probabilidad de esas diferencias.
Referencias
| Lowe, R., Shirley, N., Bleackley, M., Dolan, S., & Shafee, T. (2017). Transcriptomics technologies. PLoS computational biology, 13(5), e1005457. https://doi.org/10.1371/journal.pcbi.1005457 |