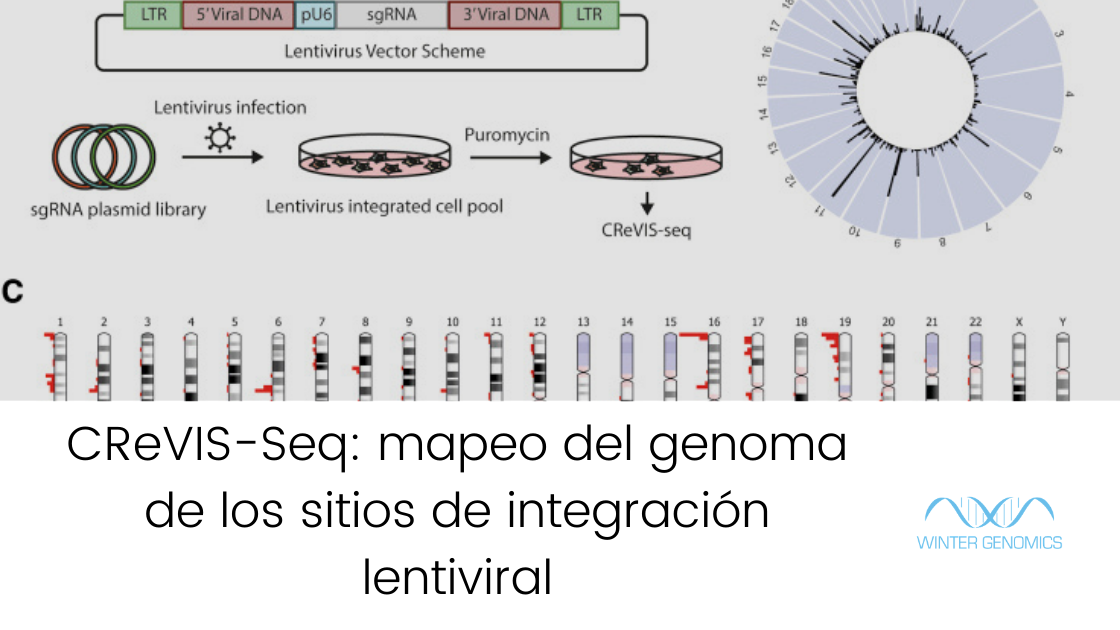
CReVIS-Seq: mapeo del genoma de los sitios de integración lentiviral
Los lentivirus se han utilizado ampliamente como medio para transferir ADN exógeno a células humanas para tratar diversas enfermedades genéticas. Los vectores lentivirales están integrados en el genoma del hospedador, pero sus sitios de integración son generalmente impredecibles, lo que puede aumentar la incertidumbre para su uso terapéutico.
Para determinar los sitios de integración viral en el genoma del huésped, se han desarrollado varios métodos basados en PCR. Sin embargo, las sensibilidades de estos métodos dependen en gran medida de las secuencias del primer, y se requiere un diseño de primers optimizado para los sitios diana individuales.
Para abordar este problema, Kim et al., 2021 desarrollaron un método alternativo para el mapeo de sitios de inserción viral en todo el genoma, llamado CReVIS-seq (secuenciación de sitios de integración viral mejorada con CRISPR).
Este método se basa en los pasos secuenciales:
- Fragmentación de los ADN genómicos
- Circularización in vitro
- Escisión de la secuencia diana en una forma específica de ARN guía de CRISPR
- Secuenciación de alto rendimiento de los fragmentos de ADN linealizados de manera no sesgada
- Identificación de la inserción viral sitios mediante análisis de secuencia.
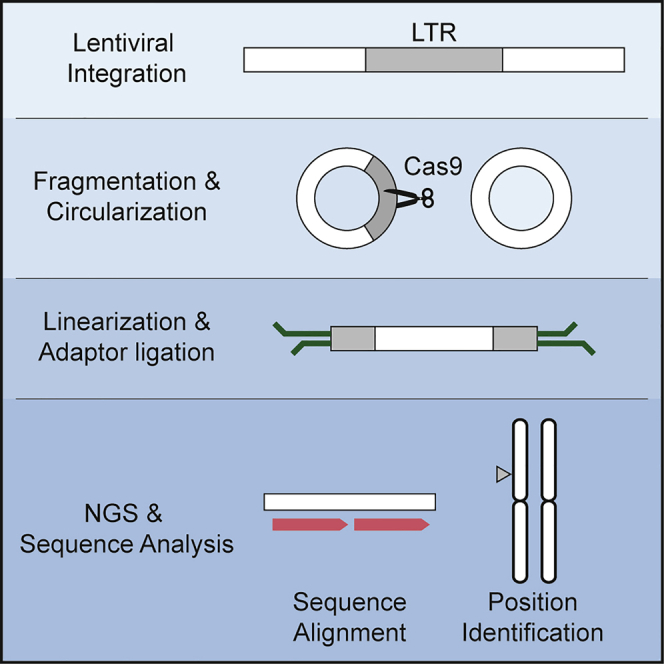
CReVIS-seq no se ve afectado por los sesgos que podrían introducirse durante el paso de enriquecimiento de la diana mediante la amplificación por PCR utilizando primers específicos del sitio. Además, encontraron que CReVIS-seq multiplexado, utilizando colecciones de diferentes RNA de guía única (sgRNA), permite la identificación simultánea de múltiples sitios diana y variaciones estructurales (es decir, genoma viral circularizado), tanto en clones de células individuales como en poblaciones de células heterogéneas.
Lee más:
https://pubmed.ncbi.nlm.nih.gov/33768124/


